Briefings in Bioinformatics | 计算机模型预测生物标记物与疾病联系!积因生物联合深圳大学发表研究论文

近日,深圳大学计算机与软件学院朱泽轩教授团队联合聚焦人工智能多组学算法领域的积因生物展开合作,双方共同完成的学术成果“Prediction of biomarker–disease associations based on graph attention network and text representation”在Briefings in Bioinformatics(IF=13.994分)期刊上发表。
该文章提出了一种新的计算模型GTGenie,这种模型能够通过图特征和文本特征预测生物标志物与疾病的关联,为候选生物标志物的快速挖掘提供可能性。本文通讯作者是深圳大学朱泽轩教授与积因生物CEO杨霄博士。

Briefings in Bioinformatics
背景介绍
生物标志物与人类疾病的关联,在复杂的病理学研究以及靶向疗法的开发上起着关键作用。
用于生物标志物发现的湿实验室具有实验成本高、费时费力等问题,运用计算机模型预测的方法则能够加快候选生物标志物的识别,从而提升效率。
结果
该文章提出了一种新计算模型——GTGenie,基于图特征和文本特征,GTGenie模型可用于预测生物标志物与疾病之间的关联。GTGenie模型运用图注意力机制来表示生物标志物和疾病之间的多种相似性,这些相似性数据来自异构信息资源。
同时,GTGenie模型通过预训练的BERT模型来学习生物医学文献中通过文本表示的生物标志物-疾病联系,将捕获的图特征和文本特征集成到双模融合网络中,以对混合实体表示进行建模。
最后,采用归纳矩阵完成法来推断缺失条目,以重建关系矩阵,并据此预测未知生物标志物与疾病的联系。在HMDD、HMDAD和lncRNADisease数据集上的实验结果表明,GTGenie模型的预测性能可以与其他最先进的方法相媲美。
方法介绍
GTGenie的模型框架如图1 所示,由三个主要部分组成,其中包括图注意力机制(Graph Attention Network, GAT)、文本关系表示(Text-based relation representation, TRR)以及双模态融合网络(Bimodal fusion network, BFN)。
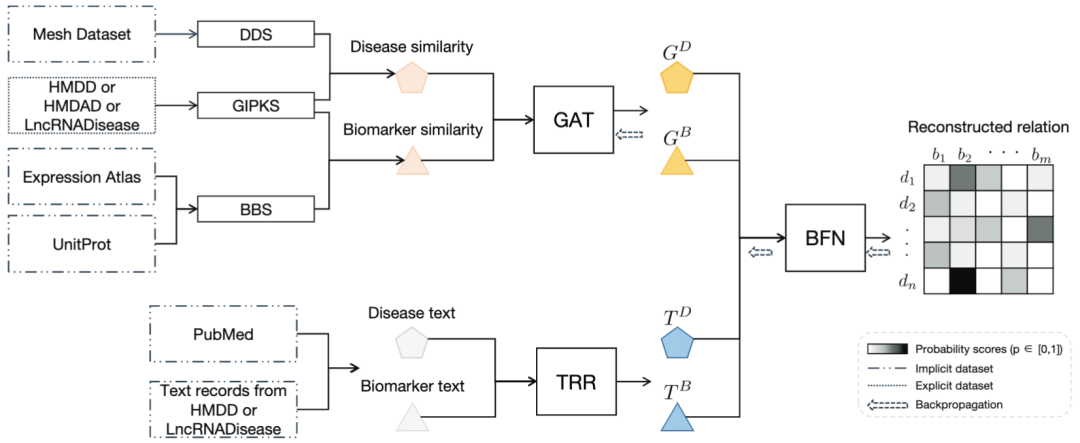
图1 :GTGenie模型框架
图注意力机制GAT
GAT的模型结构如图2所示。GAT以疾病相似性和生物标志物相似性作为输入,经过K层aggregator学习邻居之间的重要性,得到疾病和生物标志物的图特征。
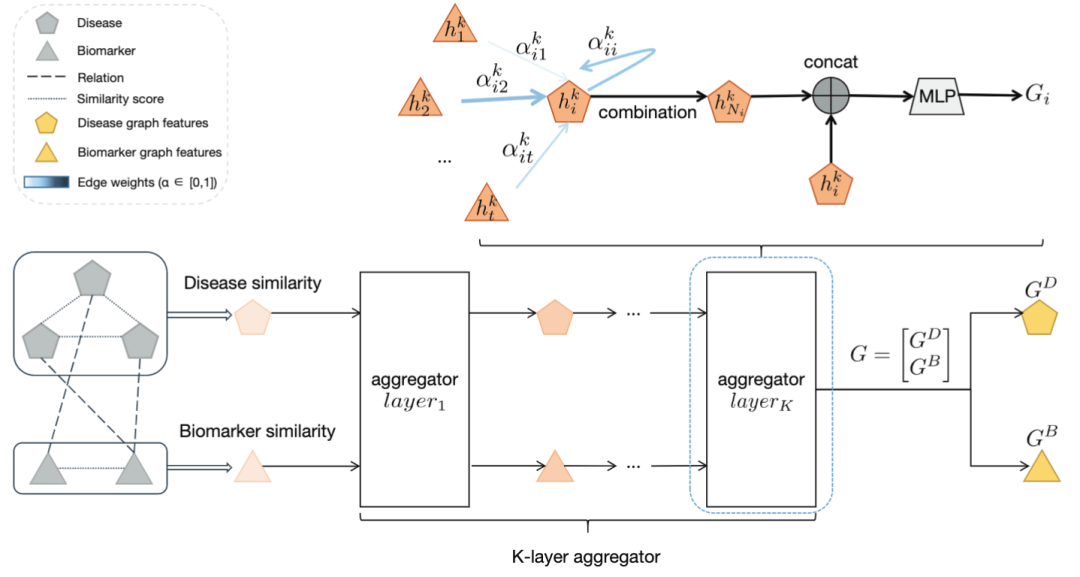
图2 :GAT模型结构
文本关系表示TRR
基于文献中检索到的相关句子,TRR能够自动测量生物标志物与疾病之间的相关性,模型结构如图3所示。首先,根据已知关系构建sub-network,每个已知关系都有一个关系句子集(ASS)对其进行描述,接着使用Biobert对ASS的每个句子进行矢量化,最后将实体所有相关的ASS embedding进行求和,得到疾病和生物标志物的文本特征。
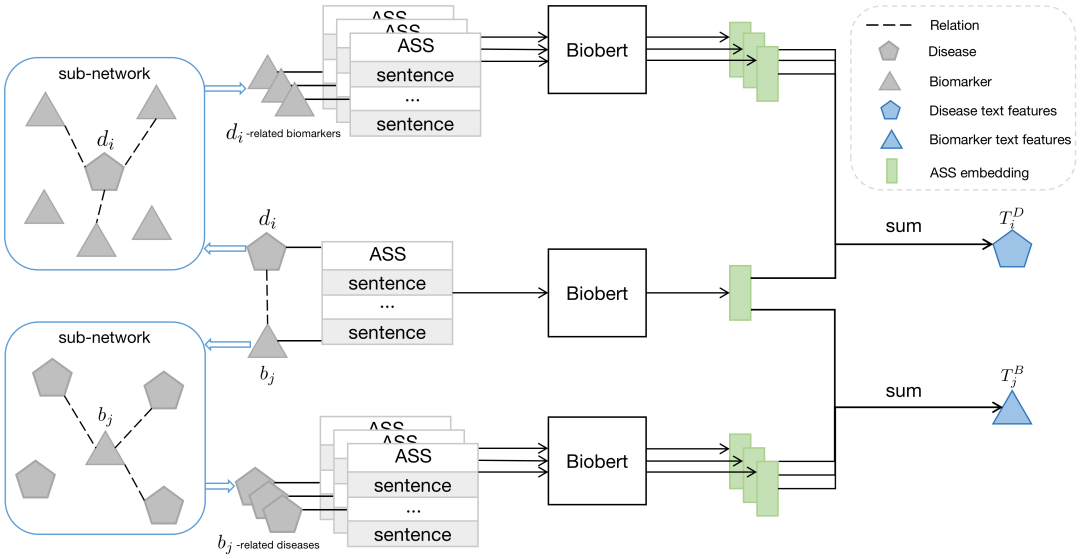
图3 :TRR模型结构
双模态融合网络BFN
BFN用于融合图特征和文本特征,并使用归纳矩阵完成技术重构关系矩阵,如图4所示。首先,将图特征和文本特征输入前馈神经网络得到精细化的表征,然后使用归纳矩阵完成技术恢复参数矩阵,获得图关系和文本关系。最后,将图关系和文本关系进行相加得到重构好的关系矩阵。

图4 :BFN模型结构
研究结果
1. GTGenie模型在 AUC结果中展示出性能优势
为了评估GTGenie模型在生物标志物-疾病关系方面的预测性能,作者使用了从HMDD v2.0、HMDAD和LncRNADisease2017数据库中收集的三个代表性数据集进行研究(AUC结果见图5)。与其他方法相比,GTGenie表现出更好的性能与泛化能力。
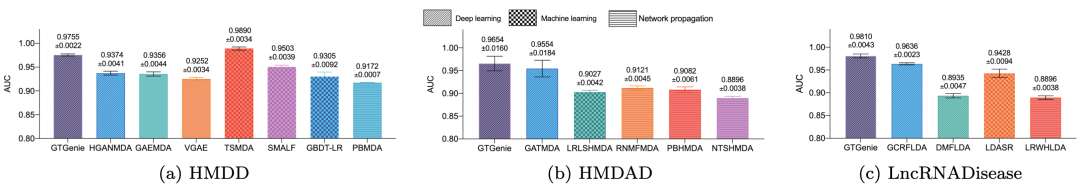
图5:不同的方法在HMDD, HMDAD 和LncRNADisease数据集的AUC结果
2. GTGenie成功验证全局Top 20生物标志物-疾病预测关系
运用其他独立数据库以及PubMed文献,作者使用GTGenie模型对数据集中未出现的top-rank预测关系进行验证。最终,全局Top 20生物标志物-疾病预测关系都被成功验证,Top 50中分别有50、40、48个关系被验证。具体验证结果见表1。
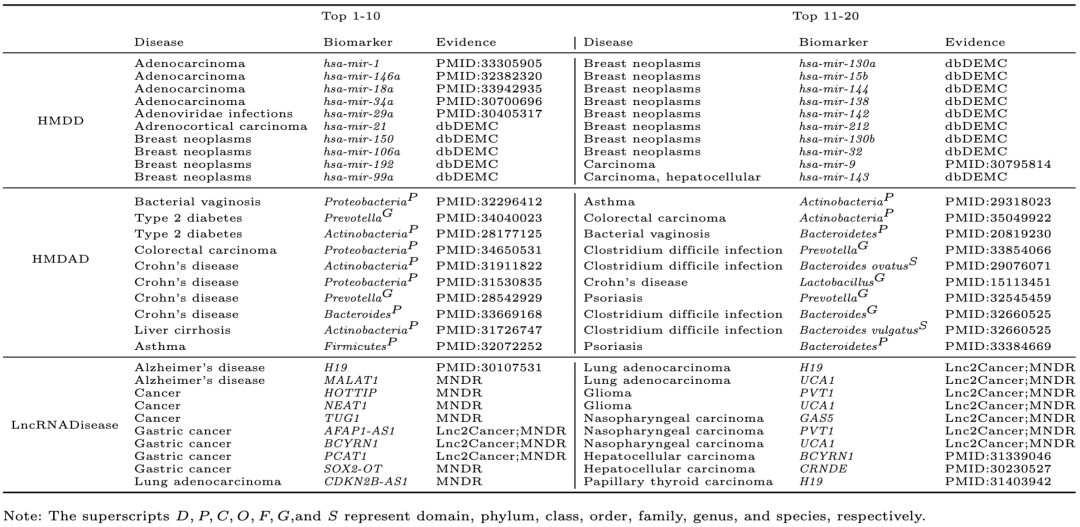
表1:全局Top 20生物标志物-疾病预测关系
3. 图特征和文本特征分别影响GTGenie模型的预测性能
与现有方法相比,GTGenie模型的创新体现在对文本特征的分析能力。图6展示了在HMDD数据集中,图特征和不同比例的文本特征对GTGenie模型结果的影响。当没有文本特征时,GTGenie模型的AUC降低了2.3%。当使用不同比例的文本特征时,GTGenie模型受到了不同程度的下降。
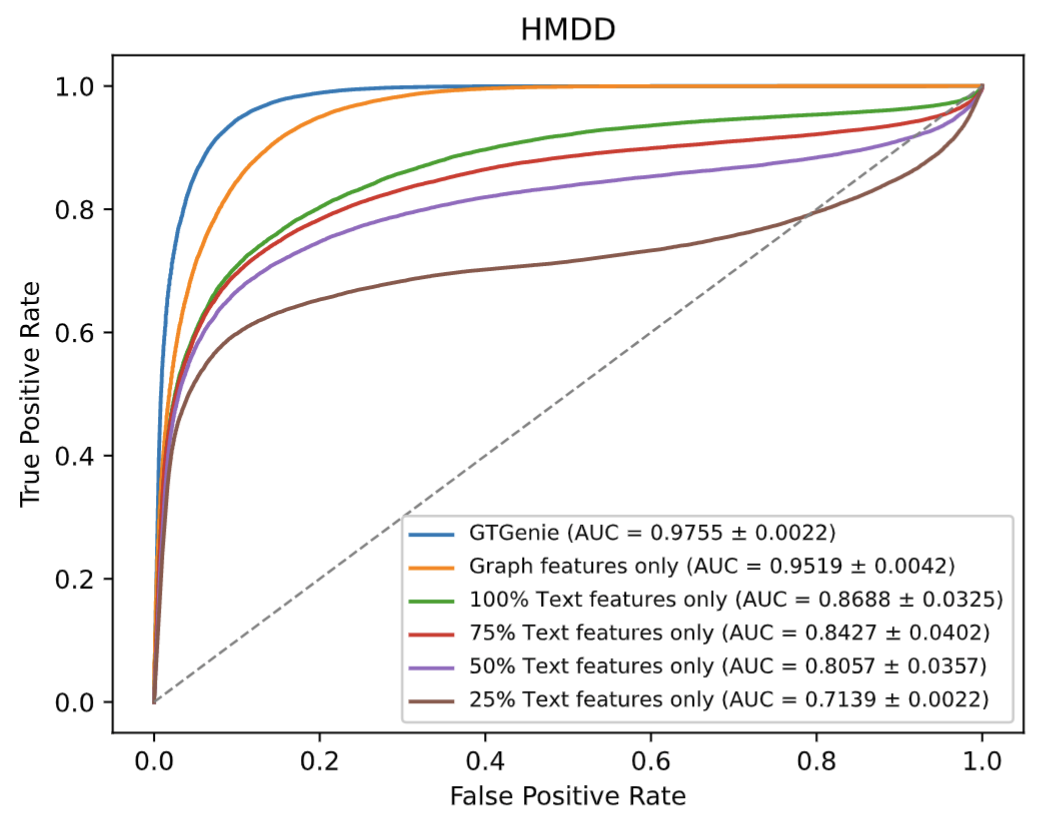
图6:图特征和不同文本特征在HMDD数据集的ROC曲线分析
研究结论
GTGenie模型整合了异构数据源和各种相似矩阵,创新性地结合图和文本特征来预测生物标志物-疾病的关系。这两种特征信息的互补有助于提高表征学习能力,达到从不同角度有效衡量生物标志物和疾病关系的效果。作者还将GTGenie模型与其他最先进的方法进行比较以及对关键组件的影响进行研究,证明了GTGenie的有效性和灵活性。
作者希望GTGenie模型能够通过融合文本特征的方式,为生物标志物-疾病的关系预测提供新见解,为加快候选生物标志物的挖掘提供更多可能。
全文链接:
https://academic.oup.com/bib/advance-article-abstract/doi/10.1093/bib/bbac298/6651308?redirectedFrom=fulltext
代码:https://github.com/Wolverinerine/GTGenie
通讯作者介绍
朱泽轩博士、教授、博导
深圳大学大数据系统计算技术国家工程实验室副主任,计算机与软件学院人工智能系系主任。主要从事生物信息学和计算智能领域的研究工作。入选斯坦福全球前2%顶尖科学家榜单(World's Top 2% Scientists 2020),广东省首批特支计划创新青年拔尖人才,深圳市首批“孔雀计划”海外高层次人才。担任中国数字音视频编解码技术标准工作组(AVS)基因压缩专题组组长,期刊IEEE Transactions on Evolutionary Computation和IEEE Transactions on Emerging Topics in Computational Intelligence副主编。已主持国家重点研发计划课题、国家自然科学基金项目多项;在IEEE Transactions on Evolutionary Computation、IEEE Transactions on Cybernetics、IIEEE Transactions on Neural Networks and Learning Systems、Bioinformatics,Briefings in Bioinformatics等期刊和国际会议发表论文150多篇,被引用5000多次。
杨霄博士
积因生物CEO,美国爱荷华州立大学生物信息学和计算生物学博士,曾任麻省理工学院和哈佛大学博德研究所计算生物学家。在分子诊断和制药领域工作十余年,曾担任Biogen、 Illumina 、Grail等公司的高级科学家 、资深软件工程师及技术负责人等职务。凭借信息学、高性能计算方面的专业知识,提供了大量大规模软件解决方案,同时在创新标志物开发、诊疗产品研发领域取得突出成绩。
积因生物
菲鹏科创旗下的广东积因生物有限公司是一家以创新分子标志物发现以及临床转化为使命的平台型研发企业。积因致力于打造多组学实验分析技术、多组学机器学习算法、自然语言处理的三大核心技术,成为全球领先的人工智能多组学平台。积因始终关注人类重大疾病,将致力于以创新技术推动医疗产业升级,最终实现洞彻生命科学奥秘,创启人类智能健康新未来的美好愿景。

